Protéine membranaire
Les protéines membranaires constituent l'une des trois principales classes de protéines à côté des protéines fibreuses et des protéines globulaires. Suivant le type de cellule et d'organite cellulaire, une membrane peut contenir des centaines de protéines différentes[1].
Chaque protéine membranaire possède une orientation définie par rapport au cytoplasme : c'est pourquoi les propriétés des faces de la membrane sont très différentes. On parle de l’asymétrie de la membrane. Dans la membrane plasmique, par exemple, les portions des protéines membranaires qui interagissent avec d'autres cellules ou avec des substances émergent dans l'espace extracellulaire, tandis que les portions qui interagissent avec des molécules cytoplasmiques sortent dans le cytosol. On peut grouper les protéines membranaires en trois classes différentes qui se distinguent par l'intimité de leurs rapports avec la bicouche lipidique.
On distingue les protéines intrinsèques, intégrées dans la membrane, et extrinsèques, associées à la membrane et les protéines ancrées aux lipides, situées à l'extérieur de la bicouche lipidique, soit à la face extracellulaire, soit à la face cytoplasmique, mais unies par covalence à une molécule de lipide située au sein de la bicouche.
Il est estimé que les protéines membranaires représentent 29,7 % du protéome[2][réf. obsolète]. Ce nombre très important reflète l'importance de cette classe de protéine, ainsi que la diversité des fonctions assurées, la plus fondamentale étant le lien entre le milieu extracellulaire et le milieu intracellulaire.
Classification des protéines membranaires
Topologies membranaires
Les protéines membranaires peuvent être classées en fonction des structures leur permettant d'interagir avec la membrane et la manière dont celles-ci s'agencent. Les types de structures mises en jeu et leurs organisations sont regroupés sous le terme de topologie membranaire.
Protéines polytopiques

1. interaction par une hélice α hydrophobe transmembranaire ;
2. interaction par plusieurs hélices α hydrophobes transmembranaires ;
3. interaction par un tonneau β transmembranaires.
Les protéines polytopiques sont des protéines en contact avec les deux compartiments définis par la membrane. Les structures en contact avec la membrane peuvent être :
- A. une ou plusieurs hélices α transmembranaires (de 1 à plus de 20 par protéine). Il s'agit de loin de la conformation transmembranaire la plus observée ;
- B. un tonneau β transmembranaire composé de 8 à 22 brins β[3].
Protéines monotopiques
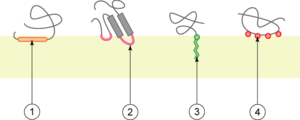
1. interaction par une hélice α amphipathique parallèle au plan de la membrane ;
2. interaction par une boucle hydrophobe ;
3. interaction par liaison covalente avec un lipide membranaire (lipidation) ;
4. interaction par liaison électrostatique directe ou indirecte (ex. : via un ion calcium) avec les lipides membranaires;
Les protéines monotopiques sont des protéines en contact avec un seul des compartiments définis par la membrane. Les structures en contact avec la membrane peuvent être :
- C. une ou plusieurs hélices α parallèles au plan de la membrane (de 1 à 3 par protéine). Ces hélices ont la particularité d'être amphipathiques ;
- D. des boucles hydrophobes ;
- E. des liaisons covalentes à un lipide, ou lipidation : il s'agit souvent de N-acylation N-terminale (liaison à un acide gras) ou de glipiation C-terminale (liaison à un glycosylphosphatidylinositol) ;
- F. des liaisons électrostatiques à des lipides. Ces liaisons peuvent se faire directement avec des phospholipides ou par l'intermédiaire d'un ion.
Classification biochimiques des protéines membranaires
Les protéines membranaires peuvent être classées en fonction de critères biochimiques, en particulier de leur difficulté à être extraites des membranes.
Protéines intégrales ou intrinsèques
Il s'agit de protéines mono- ou polytopiques interagissant fortement avec la membrane. Un détergent fort ou un solvant hydrophobe cassant la structure membranaire sont nécessaires pour les extraire.
Elle consiste premièrement à échanger l'eau avec du glycérol puis, à congeler la membrane cellulaire à grande vitesse et créer une réplique grâce à la microscopie électronique à balayage. On distinguera alors la face exoplasmique(E) et la face protoplasmique(P). On observera les protéines traversant la membrane au microscope électronique à transmission.
Les premières protéines - dont les protéines intrinsèques - ont été identifiées sur les globules rouges à l'aide de la fixation de l'isotope radioactif 125 de l'iode, fixation possible par la lactoperoxydase. Il existe cinq classes de protéines intrinsèques :
- Type I: Glycophorine ;
- Type II: Type 1 renversé ;
- Type III: band 3 ;
- Type IV: band 3 dimériques ;
- Type V.
Les protéines intrinsèques, qui pénètrent dans la bicouche lipidique. Ce sont des protéines transmembranaires, c'est-à-dire qu'elles traversent toute la bicouche lipidique et possèdent donc des domaines sortant des faces extracellulaire et cytoplasmique de la membrane. Dans certaines protéines intrinsèques, un seul segment traverse la membrane, alors que qu'il y en a plusieurs chez d'autres. Les travaux basés sur le séquençage du génome font penser que les protéines intrinsèques constituent 20-30 % de toutes les protéines codées[1].
Étude des protéines intrinsèques
Distribution des protéines intrinsèques: analyse par cryodécapage
L'idée selon laquelle des protéines peuvent traverser complètement une membrane, plutôt que de rester simplement en dehors de la bicouche , repose d'abord sur l'application d'une technique appelée réplique de cryodécapage. Dans ce procédé, on solidifie le tissu par congélation et on le frappe ensuite avec une lame, qui casse le bloc en deux fragments. À ce moment, le plan de fracture passe préférentiellement entre les deux feuillets de la bicouche lipidique. Quand les membranes sont ainsi fondues, on dépose des métaux sur les surfaces exposées de manière à obtenir une réplique ombrée qui peut être observée au microscope électronique. La réplique rappelle un peu une chaussée parsemée de gros cailloux: ce sont des particules associées à la membrane. Puisque le plan de fracture passe au centre de la bicouche, la plupart de ces particules correspondent aux protéines membranaires intrinsèques qui s'allongent au moins jusqu'à mi-chemin dans le centre lipidique. Si le plan de fracture atteint une particule, il la contourne au lieu de la casser en deux. Par conséquent, chaque protéine (particule) s'écarte avec une moitié de la membrane plasmique, laissant une cavité correspondante dans l'autre moitié. La technique de cryofracture a un grand avantage: elle permet d'étudier la microhétérogénéité de la membrane. Des différences locales dans certaines parties de la membrane se dessinent dans ces répliques et il est possible de les identifier .Par contre, les analyses biochimiques donnent des valeurs moyennes de ces différences[1].
Étude de la structure et des propriétés des protéines membranaires intrinsèques
À cause de leurs domaines transmembranaires hydrophiles, il est difficile d'isoler les protéines membranaires intrinsèques sous une forme soluble. Pour enlever ces protéines de la membrane, il faut normalement utiliser un détergent, comme le SDS, qui est un détergent (dénaturant les protéines) ionique (chargé), ou le triton X-100, détergent non ionique (non chargé) qui n’altère généralement pas la structure tertiaire de la protéine[1].
Solubilisation des protéines membranaires par les détergents
L’extrémité non polaire des molécules de détergent s'associent aux résidus non polaires de la protéine qui étaient à l'origine en contact avec les chaines d'acides gras de la bicouche lipidique. Par contre, l'extrémité polaire des molécules de détergent interagissent avec les molécules d'eau du milieu et laissent la protéine en solution.
Comme les lipides membranaires, les détergents sont amphipathiques : ils comportent en effet une extrémité polaire et une chaîne hydrocarbonnée non polaire. Cette structure permet aux détergents de substituer aux phospholipides et de stabiliser les protéines intrinsèques tout en les solubilisant dans une solution aqueuse. Quand les protéines ont été solubilisées par le détergent, on peut appliquer diverses techniques d'analyse pour déterminer leur composition en acides aminés, leur masse moléculaire, la séquence de leurs acides aminés, etc.[1].
Protéines périphériques ou extrinsèques
Il s'agit de protéines monotopiques interagissant faiblement avec la membrane, soit par liaisons électrostatiques, soit par l'intermédiaire d'autres protéines membranaires. Il n'est pas nécessaire de déstructurer la membrane pour les extraire. Une force ionique élevée ou l'emploi d'un agent chaotropique peut suffire.
En réalité, la distinction entre protéines intrinsèques et périphériques est confuse, parce que beaucoup de protéines membranaires sont formées de plusieurs polypeptides, dont certains pénètrent dans la bicouche lipidique et d'autres restent en surface.
Parmi les protéines périphériques se trouvent l'ankryne et la spectrine qui se lient de façon covalente (définitive).
Les protéines périphériques les mieux étudiées sont localisées à la face interne (cytosolique) de la membrane plasmique, où elles forment un réseau fibrillaire fonctionnant comme une « ossature » membranaire.
Ces protéines procurent aux membranes un support mécanique et servent d'ancrage aux protéines membranaires. D'autres protéines périphériques situées à la face interne de la membrane plasmique fonctionnent comme enzymes, revêtements spécialisés ou facteur de transmission de des signaux transmembranaires. Les protéines périphériques ont en général une relation dynamique avec la membrane, qui les recrute ou les libère en fonction des conditions du moment[1].
Les protéines membranaires ancrées aux lipides
On peut distinguer plusieurs sortes de protéines ancrées aux lipides. Beaucoup de protéines se trouvant à la face interne de la membrane plasmique sont fixées à la membrane par un court oligosaccharide complexe uni à une molécule de glycophosphatidylinositol encastrée dans le feuillet extérieur de la bicouche lipidique. Les protéines membranaires possédant ce type de liaison au glycophosphatidylinositol sont désignées comme protéines ancrées aux GPI. On les a découverts après avoir montré que certaines protéines membranaires pouvaient être libérées par une phospholipase qui reconnaît spécifiquement et scinde les phospholipides contenant l'inositol. La protéine cellulaire normale PrPc est une molécule unie au GPI, de même que divers récepteurs, enzymes et protéines intervenant dans l'adhérence des cellules. Un type rare d'anémie, l'hémoglobinurie nocturne paroxystique, découle d'une déficience dans la synthèse du GPI entraînant une sensibilité des érythrocytes à la lyse[1].
Rôles des protéines membranaires
Les fonctions des protéines membranaires peuvent être aussi variées que celle des protéines solubles : activités enzymatiques, fonctions structurales, moteurs moléculaires, etc. En raison de leur localisation spécifique à l'interface de deux compartiments différents, les protéines intégrales de membrane peuvent cependant avoir plusieurs types de fonctions additionnelles tout à fait spécifiques :
- le transport actif ou passif de molécules à travers la membrane. Le transport est dit passif lorsque le composé diffuse simplement d'un côté à l'autre de la membrane. Le transport est au contraire actif quand il est couplé à une utilisation d'énergie par la protéine membranaire (hydrolyse d'ATP, utilisation de la force proton-motrice). Le transport actif permet de transporter une molécule contre son gradient de concentration ;
- la réception/transmission de signaux à travers la membrane (exemples : hormones, neurotransmetteurs, etc.) ;
- le modelage et l'adhésion entre les membranes (jonctions serrées, ceintures d'adhérence, etc.) ;
- la reconnaissance intercellulaire (glycoprotéines) ;
- fixation de la matrice extracellulaire ;
- jouer un rôle enzymatique.
Les grands concepts de l'analyse protéomique
Électrophorèse bidimensionnelle et spectrométrie de masse
Dans sa version classique, la protéomique d’expression couple l’électrophorèse bidimensionnelle à la spectrométrie de masse. L’électrophorèse bidimensionnelle permet de séparer plusieurs centaines de formes protéiques, et en particulier un grand nombre de variants post-traductionnels, avec une dimension quantitative et une capacité unique de séparer des protéines entières. La spectrométrie de masse apporte quant à elle sa capacité à caractériser les protéines ainsi séparées avec force détails, comme l’identification de modifications post-traductionnelles atypiques, et bien sûr la détermination de leur site. Si cette technique a fait ses preuves, elle a aussi montré ses limites en matière de spectre d’analyse, par exemple des protéines minoritaires et membranaires, ou en matière de débit d’analyse et de miniaturisation. Ces limites provenant essentiellement de l’électrophorèse bidimensionnelle, d’autres schémas d’analyse protéomique ont été développés[4].
Chromatographie et spectrométrie de masse
La chromatographe est un terme qui recouvre une large gamme de techniques permettant le fractionnement d'un mélange de composants en solution par migration dans certains types de matrices poreuses.
Dans la technologie SELDI (surface enhanced laser desorption-ionization), ce qui est retenu sur la surface chromatograpique est analysé en spectrométrie de masse. Le principe consiste donc à déposer un échantillon complexe sur la surface chromatographique, puis à éluer partiellement cet échantillon jusqu’à ce que soit adsorbé un nombre de protéines compatibles avec la résolution du spectromètre de masse. Ce système est utilisé en analyse comparative pour analyser des marqueurs présents dans un échantillon par rapport à un autre. En variant le type de support chromatographique et le schéma d’élution partielle, il est souvent possible de mettre en évidence des marqueurs différentiels. Du fait de sa miniaturisation et de sa simplicité, cette technique est bien adaptée à l’analyse d’échantillons de petite taille comme des biopsies cliniques. Elle pâtit cependant de la difficulté à réaliser une analyse poussée des marqueurs ainsi mis en évidence, et est plutôt adaptée à l’analyse des protéines de petite taille. De plus, du fait même du principe de l’analyse, les pertes quant au nombre de marqueurs présomptifs sont considérables. Dans un autre schéma, ce sont les éluats successifs de l’échantillon complexe adsorbé sur la bille chromatographique qui sont analysés, ce qui permet une meilleure prise en compte de la complexité de l’échantillon. De plus, l’utilisation de techniques de digestion et d’analyse des peptides est facilitée, ce qui permet une caractérisation détaillée des marqueurs d’intérêt. Ces caractéristiques positives sont contrebalancées par une analyse plus laborieuse, et par le fait que nombre de protéines ne sont pas éluables des supports chromatographiques par un système compatible avec les étapes ultérieures fondées sur la spectrométrie de masse[4].
Électrophorèse en gel de polyacrylamide
Une autre technique efficace largement utilisée pour fractionner les protéines est électrophorèse. Cette méthode repose sur la faculté des molécules chargées de migrer quand elles sont soumises à un champ électrique. La séparation électrophorétique des protéines se fait d'habitude par électrophorèse en gel de polyacrylamide (PAGE) : polyacrylamide gel electrophoresis : les protéines sont poussées par un courant appliqué à un gel composé d'une molécule organique (l'acrylamide) qui forme un filtre moléculaire par des interactions croisées. Le déplacement relatif des protéines dans un gel de polyacrylamide dépend de la taille, de la forme et de la densité de charge (charges par unité de masse) de la molécule. Si la densité de charge est plus grande, la protéine est poussée avec plus de force dans le gel et sa migration est donc plus rapide. La densité de charge n'est cependant pas le seul facteur important dans la séparation par la technique PAGE ; la taille et la forme jouent également un rôle. Le polyacrylamide forme un filtre moléculaire à connexion croisée freinant les protéines qui traversent le gel. Le freinage est d'autant plus important que la protéine est plus volumineuse, et sa migration est plus lente. La forme intervient parce que les protéines globuleuses compactent se déplacent plus que les protéines fibreuses allongées de masse moléculaire comparable. La concentration du polyacrylamide utilisé pour le gel est aussi un facteur important. Plus faible est sa concentration, moins le gel forme de liaisons croisées et plus rapide est la migration d'une molécule de protéine. Un gel contenant 5 % de polyacrylamide peut être utile pour séparer des protéines de 60 à 250 kDa, tandis qu'un gel de 15 % conviendra mieux pour des protéines de 10 à 50 kDa.
On contrôle la progression de l'électrophorèse en observant la migration d'un colorant pisteur chargé qui se déplace juste devant la protéine la plus rapide. Quand ce colorant est arrivé à l'endroit souhaité, on coupe le courant et le gel est extrait du récipient. On colore normalement le gel par le bleu de Coomassie ou un sel d'argent pour localiser les protéines. Si les protéines ont été marquées par radioactivité, on peut les localiser en appliquant le gel sur un morceau de film pour rayons X et obtenir une autoradiographie ; on peut aussi découper le gel en fraction et isoler les protéines individuelles. d'un autre côté, il est possible de transférer les protéines du gel sur une membrane de nitrocellulose par une seconde électrophorèse afin d'obtenir un transfert (blot). Les protéines sont adsorbées à la surface de la membrane en gardant les positions occupées dans le gel. Dans un transfert Western (wastern blot), les protéines individuelles sont identifiées par leur interactions avec des anticorps spécifiques.
SDS-PAGE Électrophorèse en gel de polyacrylamide (PAGE) s'effectue habituellement en présence d'un détergent chargé négativement, le dodécylsulfate de sodium (SDS), qui se fixe massivement à tous les types de molécules protéiques. La répulsion électrostatique entre les molécules de SDS fixées provoque le dépliage des protéines sous une forme linéaire semblable, éliminant donc le facteur de séparation basé sur les différences de forme. Le nombre de molécules de SDS qui s'unissent à une protéine est approximativement proportionnel à la masse moléculaire de la protéine (environ 1,49 g de SDS / g de protéine). Par conséquent, chaque type de protéine quelle que soit sa taille, possède une densité de charge équivalente et est poussée avec la même force dans le gel. Cependant, à cause de la forte liaison croisée du polyacrylamide, les protéines volumineuses sont plus fortement retenues que les petites. La séparation des protéines par SDS-PAGE se base donc sur une seule propriété, leur masse moléculaire. Outre qu'elle sépare les protéines d'un mélange, le SDS-PAGE peut servir à déterminer la masse moléculaire de protéines différentes en comparant la position des bandes à celles de protéines de tailles connue[1].
Références
- Gerald Karp (trad. de l'anglais), Biologie cellulaire et moléculaire, Bruxelles/Paris, De Boeck, , 818 p. (ISBN 978-2-8041-6011-1 et 2804160114, OCLC 658731797, lire en ligne).
- (en) T.J. Stevens et I.T. Arkin, « Do more complex organisms have a greater proportion of membrane protein in their genomes? », Proteins, vol. 39, no 4, , p. 417-420
- (en) Schulz G.E. (2000) beta-Barrel membrane proteins, Curr Opin Struct Biol., vol. 266(4):761-775
- Pierre Lescuyer, « L’analyse protéomique : concepts, réalités et perspectives en thérapeutique », médecine sciences, , p. 587-592 (lire en ligne).
